03/10/2013
Les bactéries font et défont les espèces
 Les bactéries font et défont les espèces
Les bactéries font et défont les espèces
par Émilie Rauscher
(Science & Vie n° 1152, septembre 2013, pp. 106-111)
Les bactéries logées à l'intérieur de tout organisme vivant, y compris celles de la flore intestinale, influencent son évolution… Au point, parfois, de créer de nouvelles espèces. L'hypothèse avait été formulée il y a quelque temps déjà... En 1927, le microbiologiste américain Ivan Wallin notait :
"C'est une proposition pour le moins étonnante de dire que les bactéries, communément associées à la maladie, pourraient en fait être un facteur fondamental dans l'apparition de nouvelles espèces..."
Tellement étonnante, d'ailleurs, qu'elle fut rapidement oubliée, d'autant que les arguments pour la démontrer n'existaient pas... Ou plutôt pas encore. Car aujourd'hui, alors qu'ils mesurent encore à peine à quel point les bactéries sont partout – des abysses à nos cellules – ; alors qu'ils commencent à réaliser l'importance des relations ultimes et durables (symbioses) qu'elles ont tissées avec tout ce qui vit ; alors qu'ils découvrent enfin tout ce dont elles sont capables... finalement, les scientifiques en viennent à se dire qu'il y avait peut-être du vrai dans cette vieille idée folle !
Selon Bordenstein, professeur de biologie à l'université Vanderbilt (Nashville, EtatsUnis), lui, en est convaincu depuis plus de dix ans déjà : "Une nouvelle espèce pourrait bien apparaître, non seulement parce que ses gènes mutent au sein de ses propres cellules, mais aussi parce que les communautés microbiennes qu'elle héberge (dans ses intestins, etc.) changent ! " Et il a désormais de quoi étayer cette hypothèse, qui non seulement renouvelle notre compréhension de la complexe mécanique de l'évolution, mais fait des bactéries l'un des maîtres, aussi majeur que méconnu, du destin des espèces...
NOUS SOMMES TOUS DES ÉCOSYSTÈMES
Inspiré par Ivan Wallin, mais aussi par d'autres prédécesseurs visionnaires comme la biologiste américaine Lynn Margulis, qui avait perçu dès les années 1970 l'importance de la symbiose, Seth Bordenstein a patiemment réuni cas ambigus et phénomènes étranges, comme autant d'indices de l'influence des bactéries. Il mène des recherches, compile et dissèque les travaux dans lesquels des micro-organismes semblent être à l'origine d'une nouvelle espèce... Une gageure tant ce phénomène, la "spéciation", est discuté par les scientifiques : ses influences sont innombrables et ses causes multiples et difficiles à distinguer de ses conséquences.
Déjà se pose la question de ce qu'est une espèce. Cela semble simple si on regarde la chose d'un œil presbyte : de loin, les espèces se distinguent bien. De près, beaucoup moins. La faute à l'évolution et à la sélection naturelle qui poussent le vivant à changer en permanence, si bien qu'il est difficile de faire la part des choses quand une espèce se transforme ou que deux populations proches semblent s'hybrider. Par conséquent, depuis deux cent cinquante ans qu'ils travaillent dessus, les naturalistes, puis les biologistes ne se sont toujours pas accordés sur une définition claire... Retenons juste que des individus appartiennent à des espèces différentes s'ils ne peuvent pas se reproduire ensemble. Les raisons d'une telle barrière ? Soit l'acte est impossible entre eux, soit leurs hybrides sont condamnés ou stériles. Dans tous les cas, les ponts ont été coupés entre les partenaires et entre leurs espèces respectives — qui pouvaient n'en faire qu'une au départ. Incapables d'échanger des gènes, elles vont suivre chacune leur chemin évolutif et multiplier les différences. C'est ainsi qu'en partant d'un cousin de Velociraptor, on se retrouve quelques millions d'années plus tard avec des canards et des autruches. D'où viennent ces différences ? Comme le rappelle Seth Bordenstein, "les biologistes considèrent que l'apparition de nouvelles espèces est principalement dirigée par les changements qui se produisent dans nos gènes. Nous, nous démontrons que pour comprendre de façon globale comment se passe la spéciation, il faut certes considérer notre génome, mais aussi celui des micro-organismes qui sont en nous !" Pour le biologiste américain, la "cause première" de la séparation des espèces peut être, aussi, microbienne. Rappelons-nous que nous sommes de vrais écosystèmes, peuplés de nuées de bactéries. Tout comme deux populations appartenant à une même espèce peuvent être séparées physiquement par une montagne, ou génétiquement par un bouleversement chromosomique, elles peuvent l'être par ces mini-résidents !
Le chercheur a regroupé une collection inédite de cas où des bactéries ont sorti une artillerie lourde capable d'imposer cette ségrégation...
UN STRATAGÈME PARFAIT
Wolbachia est, à ce titre, exemplaire : cette bactérie infecte près de 60 % des arthropodes, l'immense groupe des insectes, acariens, araignées et crustacés. Parasite incontournable, elle s'insinue dans les cellules de son hôte et colonise la descendance des femelles. Pour favoriser sa transmission, elle réalise des prodiges.
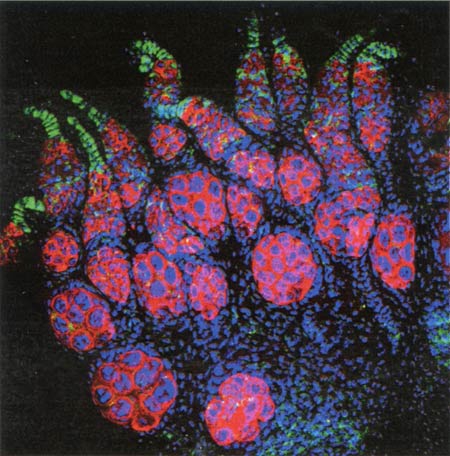
En infectant les cellules souches des organes reproducteurs de la Drosophile, la bactérie Wolbachia (en vert) modifie en sa faveur l'évolution de son hôte.
Ainsi, elle est capable d'imposer à ses hôtes une "spéciation asexuelle" : les femelles qu'elle infecte produisent sous sa houlette des ovules qui se développeront sans être fécondés par les mâles (qui deviennent ainsi inutiles). Chez des guêpes ou des acariens par exemple, on voit ainsi apparaître des lignées entièrement féminines et autonomes. "Ce cas est emblématique car immédiat, pointe Olivier Duron, spécialiste de l'évolution des symbioses à l'université de Montpellier 2. Dès l'arrivée de la bactérie, on a un isolement reproducteur, c'est-à-dire que l'individu infecté ne peut plus se reproduire avec les autres." Et qui dit isolement dit début de divergence entre les populations séparées.
Wolbachia a une seconde technique : elle sait influer sur les possibilités de reproduction de son hôte. Seules les femelles qu'elle infecte peuvent se croiser avec tous les mâles, transmettant ainsi le parasite à sa descendance. Une femelle non infectée est limitée aux mâles également non infectés, sous peine de voir ses œufs mourir. En quelques générations, près de 100 % de la population est parasitée. Ce procédé prend toute sa force quand on sait qu'il existe plusieurs types incompatibles de Wolbachia : ainsi, dans une même espèce d'arthropodes, on peut voir apparaître des groupes infectés par l'une ou l'autre souche bactérienne qui ne pourront plus se croiser !
DES ESPÈCES NAÎTRAIENT D'UN SIMPLE CHANGEMENT DE LEUR FLORE INTESTINALE
Preuve supplémentaire de la responsabilité de la bactérie : l'utilisation d'antibiotiques, en la tuant, fait tomber les barrières qu'elle dressait entre ses hôtes... À moins de s'y prendre trop tard, alors que l'évolution a déjà créé des lignées isolées. Séparées à l'origine par la bactérie, ces populations sont devenues trop différentes pour se croiser, même après sa disparition.
Ces exemples, Seth Bordenstein en connaissait l'importance, mais aussi les limites. Après tout, qu'un parasite niché au cœur même des cellules de son hôte ait un tel effet sur lui n'est sans doute pas si surprenant. Et si Wolbachia et ses consœurs sont courantes chez les arthropodes, on n'en connaît guère chez les mammifères, pour ne citer qu'eux. Ce qui limite leur impact à des groupes animaux, certes nombreux, mais bien particuliers...
Sauf que les bactéries n'ont pas besoin de s'incruster dans les cellules de leur hôte pour bouleverser sa vie. Partout dans le vivant, on observe des symbioses au cours desquelles elles se contentent de s'installer dans certains organes ! Pour rester chez les arthropodes, elles leur permettent de diversifier leur nourriture ou de s'adapter à un autre milieu, et donc de coloniser des zones interdites à leurs congénères. Grâce à elles, le termite mange du bois, le puceron conquiert de nouveaux végétaux, etc. "Puisqu'elle ouvre l'accès à une niche particulière, et donc facilite la divergence avec la population d'origine, la symbiose soutient la spéciation", souligne Olivier Duron. Voilà qui démultiplie les cas possibles !
Escherichia coli
Pourtant, Seth Bordenstein avait l'ambition d'aller encore plus loin. Pour ce défi, il reçoit en 2008 le soutien de son collègue Robert Brucker. Dans leur dernière étude, tout juste publiée, les deux biologistes soutiennent cette fois que même des bactéries banales, non symbiotiques, telles celles qui pullulent dans nos intestins, peuvent provoquer la spéciation ! "Ces dernières années, il est devenu clair que chaque animal avait des hôtes bactériens spécifiques, dont les gènes réunis (le microbiome) sont impliqués dans sa digestion comme dans son bon développement cérébral, rappelle le chercheur. Mais personne n'avait prouvé leur rôle dans la spéciation..."
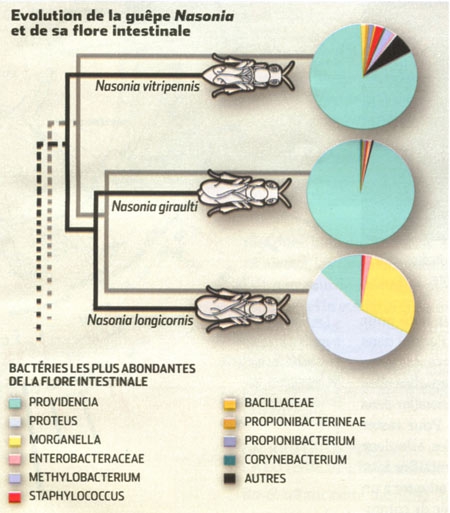
Embryon hybride de Nasonia
Leurs bactéries créent une divergence entre les guêpes qui les empêche de se reproduire. Un traitement antibiotique a mis en valeur l'influence de la flore bactérienne de 3 guêpes du genre Nasonia qui les empêche de se reproduire. Sans traitement, leurs embryons hybrides ne survivraient pas, leurs flores étant incompatibles.
Avec l'aide de trois petites guêpes du genre Nasonia, d'une bonne dose d'antibiotiques et de logique, c'est désormais chose faite. Les chercheurs savaient que lorsque ces trois espèces se croisaient, leurs hybrides étaient condamnés. Ils prédirent, en s'inspirant des expériences avec Wolbachia, qu'un traitement antibiotique aurait un effet drastique... Ils ne se sont pas trompés : près de 90 % des hybrides furent sauvés ! L'incompatibilité entre les guêpes venait donc bien des bactéries. Et réinjecter les microbes restaurait leur lourde mortalité précédente. Un scénario qui a un air de déjà-vu... Sauf que, différence fondamentale, les microbes visés étaient, cette fois, simplement ceux de la flore intestinale !
UN JOUEUR DE PLUS À LA LOTERIE DE L'ÉVOLUTION
En étudiant cette dernière de plus près, Seth Bordenstein remarqua d'ailleurs que les hybrides survivant sans traitement étaient ceux qui avaient reçu une flore proche de celle d'un de leurs parents, au lieu d'un mélange chaotique. C'est donc bien cette flore qui détermine qui vit ou meurt et, ce faisant, imperméabilise les frontières entre les espèces concernées. "Cette étude est solide et l'idée qu'elle présente est séduisante. On savait que les bactéries intracellulaires pouvaient jouer sur l'isolement de populations... Mais que les bactéries intestinales en soient également capables est nouveau ! ", s'enthousiasme Olivier Duron. Ainsi, les espèces - et l'homme ne ferait pas exception - pourraient naître et prospérer par le fait de mutations dans leur génome mais aussi, simplement, par des changements dans leur flore intestinale.
Voilà une découverte qui ajoute un joueur particulièrement riche à la grande loterie de l'évolution... Et qui dit partenaire supplémentaire dit nouveaux gènes à prendre en compte : "Notre expérience donne ainsi une bonne raison de regarder 1'hologénome, c'est-à-dire la combinaison de l'ADN de l'hôte et de ses communautés de micro-organismes résidents, souligne Seth Bordenstein. L'objet de la sélection naturelle ne serait donc pas l'individu seul, mais lui et les communautés bactériennes associées." Pour le chercheur, nous étions jusqu'ici passés à côté de la plus grande part de l'information qui constitue chaque être vivant : les bactéries, ces marionnettistes invisibles et inconscients derrière l'évolution des espèces !
Proteus vulgaris
ET CHEZ L'HOMME ?
Notre part bactérienne est plus importante qu'on ne le croit : pour dix cellules humaines, nous abritons quatorze bactéries et pour dix gènes humains, nos microbes en comptent quatorze. Outre nos quelque vingt milliers de gènes, nous en comptons donc quelque trente milliers d'origine microbienne : deux patrimoines génétiques qui doivent fonctionner "en réseau".
Le rôle des bactéries dans l'évolution des insectes décrypté, on peut se demander ce qu'il en est des mammifères ou des poissons. Seth Bordenstein et Robert Brucker se posent bien sûr la question : "Nous espérons montrer que la flore intestinale et les gènes des microbes qui la composent, le microbiome, sont importants pour la spéciation animale. Une étude récente vient d'ailleurs de montrer que l'Homme et ses plus proches parents chez les primates ont des microbiomes spécifiques à chaque espèce, qui évoluent en même temps que l'ADN nucléaire (dans le noyau des cellules)." Cette coévolution plaide pour un lien fort entre notre génome et celui de notre flore. Les études se multiplient d'ailleurs pour recenser les bactéries que nous hébergeons et mieux comprendre le microbiome riche de plusieurs millions de gènes qu'elles constituent. On sait déjà qu'il existe trois "groupes bactériens" intestinaux, comme il y a des groupes sanguins, et que les perturber a des conséquences sur notre santé.
20:16 Publié dans Biologie, Médecine | Tags : spéciation, génomique, bactéries, flore bactérienne, microbiome | Lien permanent | Commentaires (0) | ![]() Facebook | | |
Facebook | | |
20/05/2012
Médecine et génomique à l'Institut Pasteur
 Médecine et génomique à l'Institut Pasteur
Médecine et génomique à l'Institut Pasteur
Nous ne sommes pas tous égaux devant les maladies
"Tous les hommes sont égaux, mais certains sont plus égaux que d'autres" écrivait Georges Orwell. Ceci est un peu vrai face à la maladie. Si certaines pathologies sont provoquées par des mutations dans un gène unique, la plupart des maladies courantes ont à la fois une composante environnementale (nutrition, activité physique, etc.) et génétique – en témoignent les questions de nos médecins sur nos antécédents familiaux. Et qu'il s'agisse de diabète, d'asthme, de maladie d'Alzheimer, de cancers ou de maladies infectieuses, de multiples variations dans de multiples gènes interviennent.

Des facteurs génétiques de prédisposition ou de protection
Certaines nous rendent plus vulnérables face à une maladie donnée – elles touchent des gènes "de prédisposition" – d'autres au contraire nous en protègent. Une mutation dans les gènes BRCA1 ou BRCA2 confère par exemple aux femmes qui la portent (2 sur 1 000) un risque de 40 à 80 % selon l'âge de développer un cancer du sein. Les rares individus porteurs d'une mutation du gène CCR5 sont eux résistants à l'infection par le virus du sida. D'autres gènes conditionnent la réponse aux traitements. Ainsi, 11 % des patients réagissent mal à une molécule, la mercaptopurine, utilisée pour traiter certaines leucémies. Chez eux, une activité réduite de l'enzyme TMPT freine l'élimination du médicament, induisant des effets très toxiques. Un test génétique est donc désormais recommandé aux États-Unis afin d'éviter ce traitement ou de rappliquer à très faibles doses chez ces patients.
Pour appréhender l'ampleur des recherches sur ces gènes de sensibilité aux maladies ou aux traitements et les perspectives médicales qui en découlent, il faut revenir sur la fantastique épopée scientifique qui a marqué le début du XXIe siècle.

Naissance de la génomique
Tout a commencé par l'aboutissement d'un projet titanesque, qui dura 13 ans : le Projet Génome Humain. Achevé en 2003, il révélait l'intégralité du texte d'un ADN humain – sa "séquence" – soit 3 milliards de lettres[1].
Il ouvrait l'ère de la génomique, rendant possible l'exploration des quelque 20 500 gènes contenus dans le génome humain, facilitée d'année en année par l'automatisation des techniques de séquençage et les progrès de la bio-informatique. Il a montré qu'il y a très peu de différences génétiques entre les individus ou les populations et pas de différences majeures entre groupes ethniques, abolissant définitivement le concept de race. Les génomes de deux individus pris au hasard dans la population mondiale ont en moyenne à peine 0,1 % de différences. Mais sur les trois milliards de lettres du texte de l'ADN, ce 0,1 % correspond tout de même à 3 millions de variations... dont bon nombre expliquent nos différences de vulnérabilité face aux maladies.
Nos différences génétiques cartographiées
Comment les étudier ? Dans la foulée du projet Génome Humain, un autre grand programme international – Hap-Map –, a dressé un catalogue des différences et des similarités génétiques entre individus. À partir des génomes de 270 individus d'origines africaine, asiatique et européenne, les variations génétiques les plus fréquentes (des changements ponctuels d'une lettre dans le texte de l'ADN, présents chez plus de 5 % des individus) ont été répertoriées. Trois millions de telles variations ont été cartographiées. Ces "balises" dans l'ADN ont permis de vastes études d'association sur des génomes entiers, portant chacune sur des centaines voire des milliers d'individus : les GWAS (genome-wide association studies). Schématiquement, un panel d'individus sains est comparé à un panel d'individus touchés par une maladie donnée, sur la base du catalogue Hap-Map : si certaines variations génomiques sont beaucoup plus fréquentes chez les malades, elles sont associées à la pathologie. En janvier 2012, plus de 1000 "GWAS" avaient été publiées, mettant en évidence plusieurs centaines de nouveaux facteurs génétiques de risque pour des maladies courantes : diabète, maladies cardio-vasculaires, maladies neuro-dégénératives (Alzheimer, Parkinson), cancers, maladies mentales comme la schizophrénie ou l'autisme (voir encadré), maladies infectieuses (hépatites B, C, sida, tuberculose…).

Le projet "1000 génomes"
Ainsi, plus d'axes de recherche totalement inattendus et de pistes thérapeutiques nouvelles ont été identifiés depuis l'an 2000 qu'au cours du XXe siècle, et révolution de la génomique ne cesse d'apporter des outils toujours plus précis pour les recherches sur notre vulnérabilité aux maladies. Le projet "1000 génomes" lancé en 2008 vise désormais à fournir un grand nombre de séquences complètes de génomes humains à partir de toutes les populations du globe. Mais aussi à répertorier dans ces génomes des millions d'autres variations, dites "rares" (présentes chez moins de 0,5 % de la population), et divers "accidents" dans l'ADN qui n'ont pas à ce jour été cartographiés, comme de petites portions manquantes (délétions) ou surnuméraires (insertions). "Les chercheurs qui ont trouvé des régions du génome associées à une maladie pourront regarder ces données et avoir un panorama presque complet des variations génétiques dans ces régions pouvant contribuer directement à la maladie", souligne un des auteurs du projet. À ce jour, 2 500 échantillons d'ADN provenant de 27 populations à travers le monde doivent être analysés.
Vers une médecine prédictive et personnalisée ?
Côté applications, la révolution médicale annoncée au début des années 2000, qui permettrait de prédire à chacun quelles maladies il risque de développer et de proposer des traitements à la carte en fonction du profil génétique, n'en est qu'à ses prémices. Mais pour beaucoup, il ne s'agit plus de savoir si elle surviendra, mais quand. Un des freins tient au fait que l'on s'attendait à découvrir quelques gènes majeurs associés aux maladies, et qu'on en a trouvé des dizaines, voire des centaines : en août dernier, une équipe internationale annonçait avoir découvert 29 nouveaux variants génétiques associés à la sclérose en plaques, s'ajoutant à 23 variants déjà connus... soit une cinquantaine identifiés pour cette seule maladie ! Il reste par ailleurs difficile d'évaluer parmi les gènes de "prédisposition" à une maladie lesquels sont les plus "prédictifs" d'un risque. Aussi, les sociétés qui proposent aujourd'hui sur Internet des tests génétiques révélant nos risques de maladies à partir d'un échantillon de salive sont-elles montrées du doigt pour leur manque de fiabilité... Au fil des avancées, une médecine prédictive plus fiable verra cependant le jour avec à la clé des mesures de prévention mieux ciblées. L'avancée des connaissances en pharmacogénomique, l'étude des facteurs génétiques individuels en jeu dans la réponse aux traitements, en plein développement, conduira parallèlement à l'essor de la médecine personnalisée : déjà, plusieurs traitements existent accompagnés de tests génétiques "compagnons", qui conditionnent leur utilisation chez un patient selon qu'il est "bon répondeur" ou bien à risque d'effets indésirables.

1 000 dollars pour séquencer un génome
Un élément de taille est venu récemment conforter cette tendance à envisager une médecine du futur tenant compte du profil génétique du patient : la chute drastique des coûts du séquençage. Si en 2001 des centaines de millions de dollars étaient nécessaires pour obtenir la séquence génomique complète d'un individu, ce prix tombait à 50 000 dollars en 2009. En février 2011, une société américaine annonçait dans la revue scientifique Nature réduire ce coût à 1000 dollars. Le séquençage d'un génome serait bientôt à un prix si abordable qu'il pourrait être généralisé à tout un chacun. À quand notre séquence génomique dans notre carte Vitale ? Les questions éthiques et sociétales soulevées par cette perspective – la protection des données génétiques vis-à-vis des assurances ou des employeurs par exemple – deviennent urgentes à résoudre...
En attendant, chaque variation génétique associée à une maladie est une piste nouvelle pour les chercheurs. Il faut alors comprendre le mécanisme biologique dans lequel le gène impliqué intervient, trouver le moyen d'agir en conséquence pour traiter la maladie... La décennie précédente a fourni une avalanche de données génétiques associées aux maladies : seul le sommet de cet iceberg d'informations a été exploré. Son exploitation fournira sans doute bien des solutions inédites face aux maladies qui nous menacent.
Génomique et paludisme : vers de nouvelles pistes de recherche
Près d'un million de morts et 500 millions de cas par an, des traitements de moins en moins efficaces : le paludisme, un des premiers fléaux sanitaires de la planète, est une véritable urgence pour la recherche et l'étude des facteurs de sensibilité à cette infection apporte l'espoir de nouvelles voies d'investigation.
À l'Institut Pasteur, l'unité de Génétique fonctionnelle des maladies infectieuses, dirigée par Anavaj Sakuntabhai, avait déjà contribué à la découverte d'une mutation génétique conférant une résistance au paludisme chez 18 à 25 % des individus en Asie du Sud-Est, après une étude sur 3 500 personnes en Thaïlande. "Cette mutation est située dans un gène impliqué dans le stress oxydatif : ce phénomène intervient dans le vieillissement et la destruction des globules rouges, lieux de multiplication des parasites", explique Jean-François Bureau, un des chercheurs de l'unité.
Aujourd'hui, l'équipe pasteurienne lance une vaste étude de recherche de gènes associés à l'infection (GWAS[2]). Sa source : les échantillons d'ADN de plus d'un millier d'habitants des villages de Dieimo et de Ndiop au Sénégal – suivis depuis 1990 par l'Institut Pasteur de Dakar et l'Institut de Recherche pour le Développement (IRD) –, et de 600 personnes au Pérou. "Nous allons cibler nos recherches en définissant des sous-groupes dans ces populations exposées au paludisme : en fonction du nombre d'accès palustres, de leur sévérité, de l'âge – tes enfants étant les plus touchés –, etc." précise Richard Paul, autre chercheur de l'équipe. "Grâce au suivi de ces habitants depuis 20 ans, nous disposons de quantité de données, parfois depuis la naissance. À chaque fois que nous découvrirons une mutation dans un gène, nous saurons ainsi à quel type de cas clinique elle est associée." Les premiers résultats devraient être disponibles dans un an.
Autisme : la part des gènes
Dès les années 70, des études chez les jumeaux mettaient en évidence une composante génétique dans l'autisme, trouble envahissant du développement qui touche un enfant sur 100. En 2003, les premiers gènes associés à des cas d'autisme et de syndrome d'Asperger (autisme avec langage et sans retard mental) étaient découverts à l'Institut Pasteur dans l'unité de Génétique humaine et Fonctions cognitives dirigée par Thomas Bourgeron. Aujourd'hui, plus de cent gènes sont associés à la maladie. Quelle part jouent-ils ?
"C'est très complexe", explique Thomas Bourgeron. "D'abord, il n'y a pas "un" autisme, mais un spectre de troubles autistiques, donc quantité de patients différents et autant de cas particuliers. Ensuite, l'impact de chaque gène diffère. Par exemple, la présence de mutations dans le gène SHANK3 que nous avons découvert signe quasiment toujours un autisme.
Ces cas-là sont "très génétiques". Chez d'autres patients, les mutations identifiées sur d'autres gènes n'expliquent pas à elles seules la maladie, qui relève probablement d'autres facteurs génétiques non identifiés et peut-être aussi de facteurs épigénétiques et environnementaux. Quoiqu'il en soit, ces gènes de vulnérabilité sont autant de pistes de recherche pour mieux comprendre les autismes." Dans le laboratoire de l'Institut Pasteur, généticiens côtoient neurobiologistes et psychiatres. Il ne s'agit pas seulement d'isoler des gènes associés à l'autisme mais de comprendre à quoi ils servent. "Pour définir la fonction de gènes récemment Impliqués, nous cultivons actuellement des neurones avec des cellules exprimant ces gènes mutés" explique Thomas Bourgeron. "Nos résultats préliminaires montrent des différences dans la taille des neurites, ces prolongements neuronaux pouvant donner de nouvelles connexions".
D'autres gènes impliqués interviennent dans le fonctionnement des points de communication entre les neurones, les synapses. Des images en 3D de cerveaux d'enfants autistes, reconstituées à partir d'IRM, sont aussi analysées en vue de relier d'éventuelles modifications aux mutations génétiques trouvées chez ces patients. C'est donc une cascade d'études qui découle des découvertes de gènes de vulnérabilité, avec à la clé l'espoir d'améliorer la prise en charge des enfants touchés.
Source : La lettre de l'Institut Pasteur, mai 2012.
15:04 Publié dans Biologie, Médecine | Tags : médecine, génomique | Lien permanent | Commentaires (0) | ![]() Facebook | | |
Facebook | | |


